Ergebnisse
Simulation von Proteinlösungen
In ersten Experimenten wurde versucht, Proteinlösungen in Simulationen möglichst realistisch darzustellen. Dazu wurden als Modellproteine Lactamasen gewählt und jeweils 8 Proteine in unterschiedlich großen Wasserboxen simuliert.
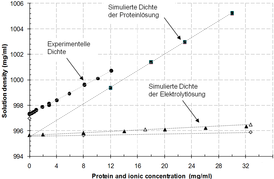
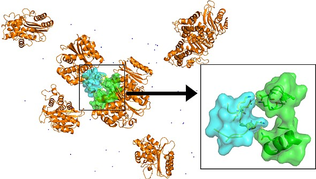
Die Simulationsboxen umfassten 302000 Atome und beinhalteten jeweils 8 Lactamasen sowie Wasser als Lösungsmittel. Die Proteinkonzentration betrug in dieser Box 110 mg/ml, die Dichte der Proteinlösung lag bei 1,004 g/ml. Zur Bestimmung der Dichte wurde eine verlässliche Methode entwickelt. In weiteren Schritten wurden die Simulationsboxen bei gleichbleibender Proteinanzahl vergrößert, um experimentell realistische Proteinkonzentrationen zu erreichen (Abb. 1). Die untersuchten Proteinkontakte (Abb.2) zeigten, dass lediglich in großen Systemen mit mehr als 250A Boxlänge unabhängige Proteinbewegungen stattfanden.
|
|
|
Hydration von Proteinen
|
|
In diesem Teilprojekt wurden Gasphasen-Simulationen von Candida antarctica Lipase in einem Trägergas durchgeführt. Dabei wurden unterschiedliche Mengen an Wassermolekülen zugegeben und deren Einfluss auf das Protein untersucht. Die Simulationen wurden durch experimentelle Gasphasen-Reaktor-Experimente validiert.
Alle Simulationen wurden mit CALB als Modellprotein und unter Kontrolle der Wasseraktivität durchgeführt. Dabei zeigte sich, dass die Bildung von Wassernetzwerken stark von der thermodynamischen Wasseraktivität abhängt, was durch einen Kooperationspartner experimentell bestätigt werden konnte [1]. Die Simulationen und auch Experimente wurden mit Hilfe eines Trägergases (Argon) durchgeführt. Bei niedrigen Wasseraktivitäten zeigte sich ein gezieltes Andocken an Wasserbindungsstellen an der Proteinoberfläche (Abb. 3). Bei einer Aktivität von aw=0.5 formte sich ein stabiles Netzwerk mit 311 Wassermolekülen an der Oberfläche der Lipase. Höhere Wasseraktivitäten führten zu dickeren Wasserschichten am Protein, welche ab ca. 1600 Molekülen instabil wurden und in Abspaltung von Tröpfchen resultierte. Zudem zeigten die Experimente, dass die Substrat-Bindetasche eine deutlich erhöhte Flexibilität aufwies, wohingegen die Gesamtflexibilität von CALB keine Änderung zeigte.