Beschreibung
|
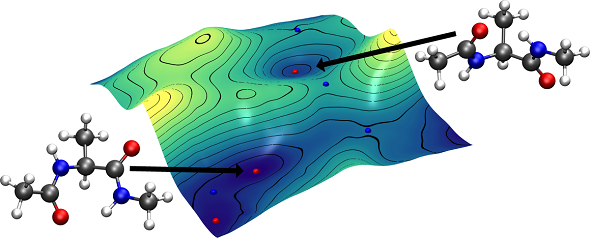
Bei einer chemischen Umsetzung ist der Pfad kleinster freier Energie (Minimum-Free-Energy-Path, MFEP) der Reaktionspfad mit dem höchsten statistischen Gewicht. Er gibt Aufschluss darüber, welche Atome an einer Reaktion beteiligt sind. Aus dem MFEP können die Änderung der freien Energie bei der Reaktion sowie die Energiebarriere der Reaktion bestimmt werden. Erstere ist beispielsweise die freie Bindungsenergie eines Liganden an einen Rezeptor. Aus letzterer kann die Reaktionsrate ermittelt werden. So können Umsetzungen untersucht werden, die um viele Größenordnungen langsamer ablaufen als mit Molekulardynamik direkt zugängliche Prozesse.
In diesem Projekt wird eine Methode entwickelt, um den MFEP mit Hilfe von Umbrella-Sampling Simulationen zu lokalisieren. Umbrella-Integration [1] wird verwendet, um die ersten beiden Ableitungen der freien Energie nach Atomkoordinaten aus den Ergebnissen von Molekulardynamiksimulationen zu erhalten. Diese Ableitungen, an einigen Punkten entlang eines Test-Pfades ausgewertet, erlauben es, den Test-Pfad zum MFEP zu konvergieren. Traditionell war nur die erste Ableitung zugänglich, was Geometrieoptimierungsverfahren auf lineare Optimierer beschränkt. Mit Umbrella Integration kann auch die zweite Ableitung berechnet werden. Das wird die Konvergenz der Pfadoptimierung erheblich verbessern.
Die Methode ist intrinsisch parallel: an allen Bildern entlang des Pfades (typischer Weise 20–100) müssen unabhängig voneinander Molekulardynamiksimulationen durchgeführt werden. Diese werden selbst wieder parallel ausgeführt. Das erlaubt die effiziente Nutzung von modernen Rechenclustern mit mehreren tausend Prozessoren.
[1] J. Kästner, W. Thiel, J. Chem. Phys. 123, 144104 (2005)
- Ergebnisse
- Publikationen
- Startseite des Projekts